蛋白核酸互作是指蛋白和DNA或者RNA之间的相互作用。主要的互作类型有蛋白和蛋白、蛋白和DNA、蛋白和RNA、RNA和小RNA之间的互作模式。作为中心法则的三大成员,他们之间的互作关系以及调控关系是后基因组时代重要的研究领域,在表观遗传学等不同研究方向上也具有重要的研究意义。而在实际研究过程中所使用到的蛋白核酸互作的实验技术不一,本期将从原理、应用场景和实验设计和大家重点分享几个蛋白核酸互作技术:CO-IP(蛋白-蛋白)、ChIP-seq/PCR(蛋白- DNA)、CHIRP(RNA-蛋白-DNA)、RIP-seq/PCR(蛋白-RNA)、RNA pulldown-WB/质谱(RNA-蛋白)、Luciferase(转录因子-启动子或3‘UTR- microRNA等),旨在帮助大家更快的将这些技术应用到科学研究中。
实验原理
CO-IP免疫共沉淀,是研究蛋白和蛋白互作的一种技术,其原理是利用目的蛋白的抗体从样本中捕获和富集与其存在相互作用的蛋白或者蛋白复合体的技术,是比较经典的研究蛋白互作的技术之一(往期研究蛋白互作的文章)。抗体富集后的样本可以直接使用WB技术检测待验证的互作蛋白,也可将富集后的样本进行质谱(Mass Spectrum)和生信分析,获得更多未知互作蛋白的信息。

CO-IP流程示意图
应用场景和实验设计
该技术主要应用于研究已知蛋白和已知或者未知蛋白互作的探究,是研究蛋白和蛋白互作的超实用工具之一。
样本类型:一般是细胞裂解液,可以是外源过表达目的蛋白(加标签),也可以使用内源蛋白抗体直接做IP。另外细胞可以是所研究的肿瘤细胞(内源验证),也可以使用HEK293T做外源验证。
实验分组:空细胞(可选)、IgG(以排除抗体的非特异性吸附)、目的抗体(富集目的蛋白)。
结果说明:
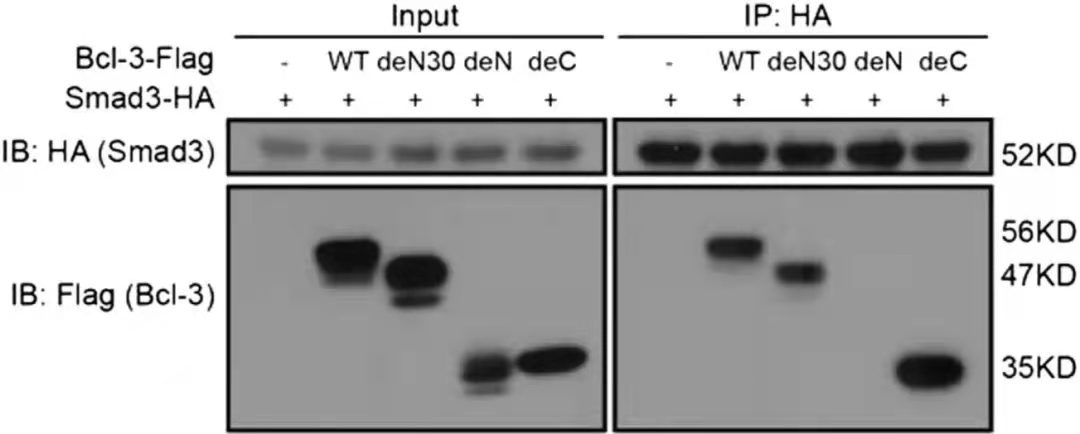
CO-IP研究蛋白互作结果[1]
图中WT: 1–446 amino acids; deN30: 31–446 amino acids; deN:125–446 amino acids; deC:1–330 amino acids
CO-IP结果中的Input组一方面是为了衡量上样量的一致性,一方面可以检测细胞裂解中目的蛋白是否可被检测到以及相应的条带大小。IP组的结果用以说明目的蛋白是否和下游蛋白存在互作。该实验通过构建不同截短的Bcl-3-flag探究和Smad3-HA的互作结构域,结果表明Bcl-3的N端序列(de N组)的31-125氨基酸区域是和Smad结合所必须的。
实验原理

ChIP实验流程示意图[2]
应用场景和实验设计
样本类型:细胞或组织
实验分组:ChIP实验的对照选择比较关键,是评估实验结果可靠性的重要依据,一般设有如下分组:
① 实验内对照:即Input DNA(断裂后的基因组DNA)作为ChIP实验的内对照。内对照该组可验证实验体系中染色质的断裂效果;若根据Input中的DNA靶序列的含量和IP后样本中的靶序列含量,可间接推算ChIP的实验效率;
② 阳性抗体对照:常用组蛋白抗体作为阳性对照。通常选择与已知序列相结合的、各物种之间比较保守的蛋白抗体;
③ 阴性抗体对照:常用IgG作为阴性对照组,也可以选择目的蛋白抗体宿主的血清蛋白。
结果说明:ChIP实验结果中最关键的结果是相对免疫沉淀的效率(富集效率)和相对的蛋白与DNA 结合能力(富集倍数),该指标和靶蛋白在细胞中的丰度高度相关,一般来说可以将相对于IgG对照组的3-5倍定为实验质控的最低阈值,同时可根据已报导文献做相应的调整。
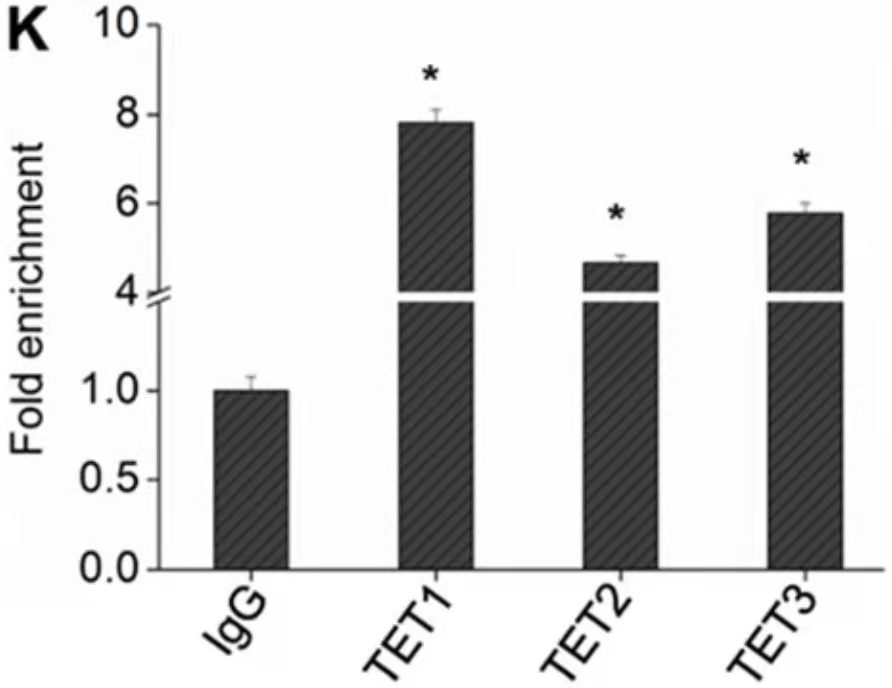
ChIP- PCR结果展示[3]
该实验通过3个目的蛋白的抗体进行ChIP(anti-TET1, anti-TET2, anti-TET3),然后qPCR鉴定转录因子和启动子的结合,结果表明,TET1/2/3均可以和PTENp1的启动子区结合,其中TET1的结合更强。
实验原理
CHIRP是一种检测体内与RNA绑定的DNA和蛋白相互作用的方法。该技术是通过设计与目标RNA序列反向互补的生物素探针,把目标RNA拉下来以后(此步骤类似于RNA pulldown),则与其共同作用的DNA染色体片段就会被间接富集到链霉亲和素磁珠上,最后通过qRT-PCR或测序来测定目的RNA、DNA序列,也可通过Western或质谱分析来测定蛋白所富集的蛋白种类。
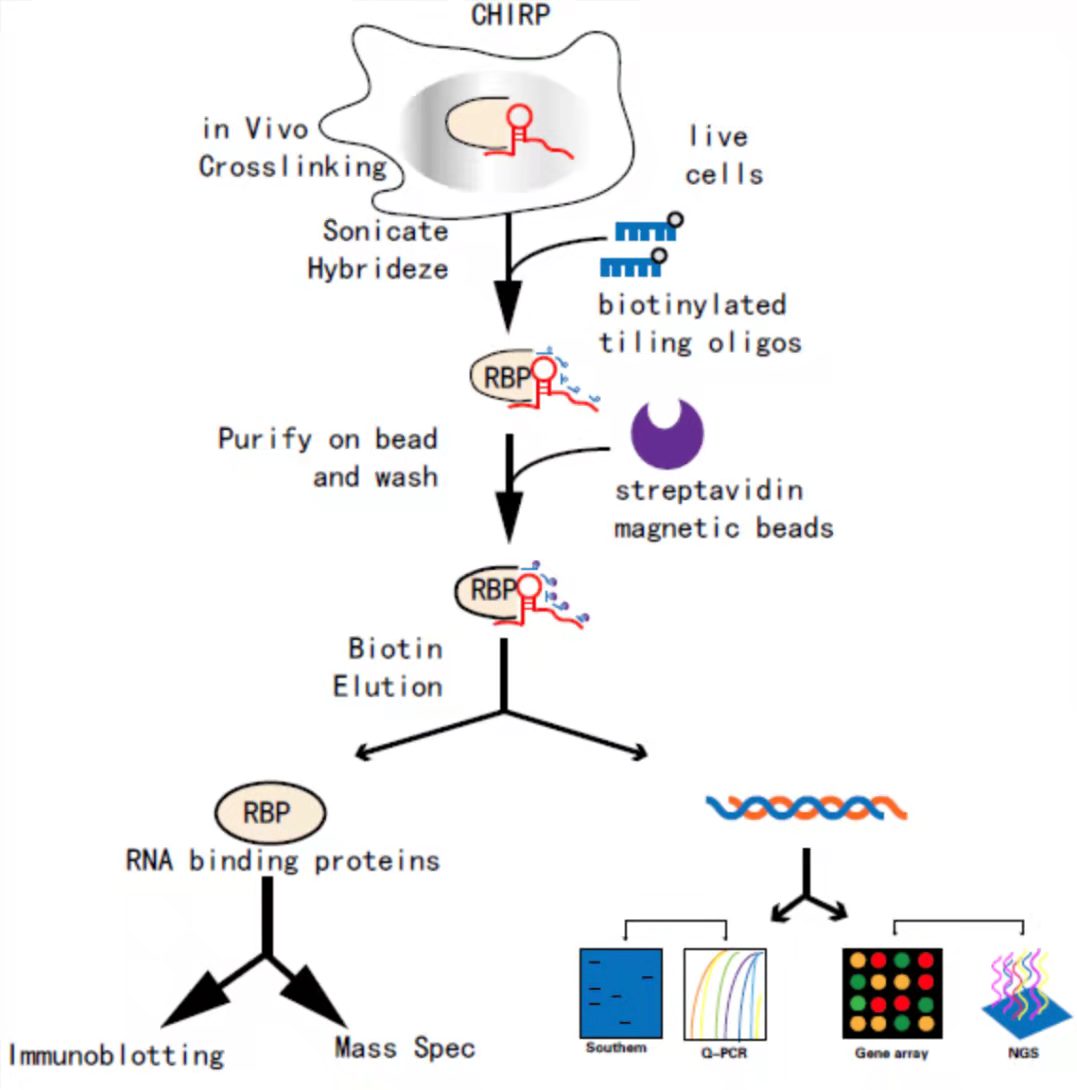
CHIRP实验流程图
应用场景和实验设计
该技术最大的优势在于既可研究RNA、蛋白质和DNA形成的复合体之间的互作,也可研究它们之间的两两互作;利用CHIRP-MS实验寻找与目的lncRNA结合的蛋白(CHIRP-Protein),可不受lncRNA长度的限制;利用CHIRP实验可研究circRNA与蛋白质(CHIRP-Protein)或DNA(CHIRP-DNA)之间的互作。
样本类型:一般为细胞。
实验分组:
IP组:即实验组,用于鉴定lncRNA/circRNA作用于基因组的位置;
lacZ组:外参对照组,用于证明ChIRP探针的特异性;
Input组:捕获前分离提取的基因组DNA作为内参对照组,证明探针特异性;
Positive组:阳性对照组,通过已知验证有效的探针,通过WB检测已知结合蛋白质,证明整个ChIRP实验体系的有效性;目标lncRNA/circRNA qPCR:证明ChIRP探针对目标lncRNA的正确结合及有效捕获。
实验原理
和ChIP不同的是,RIP是研究蛋白和RNA互作关系的一种技术手段。其原理和CO-IP以及ChIP类似,也是基于抗原-抗体的方式,利用目的蛋白抗体富集和沉淀下来蛋白所结合的RNA的过程。流程如下:裂解细胞-抗体孵育-免疫沉淀- RNA纯化-建库测序/qPCR。
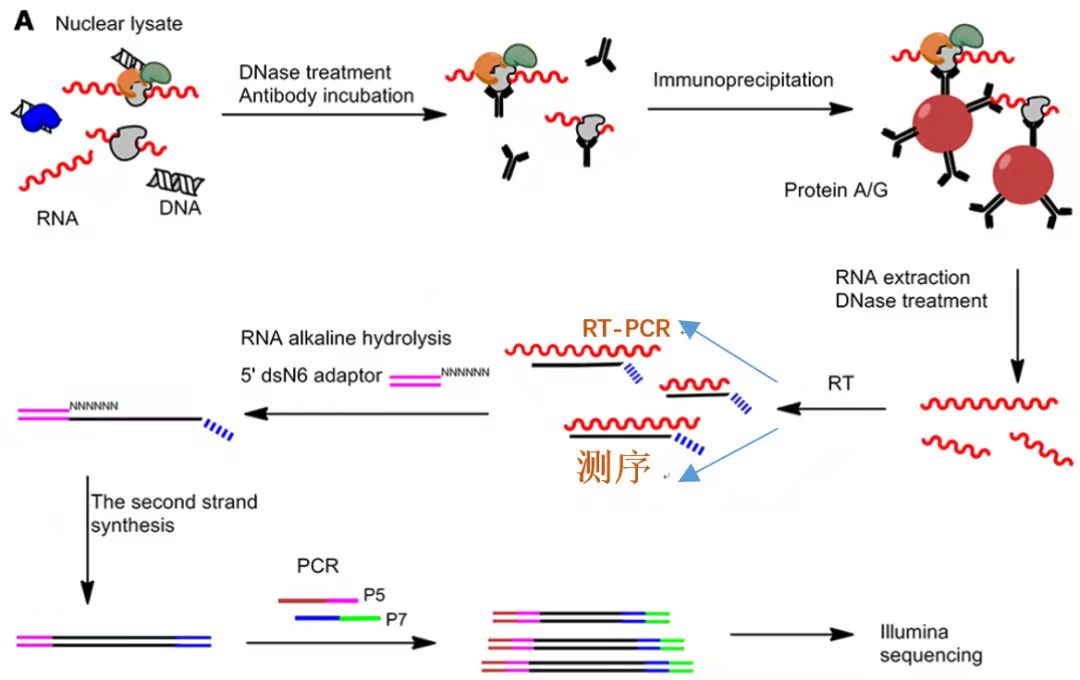
RIP实验流程图示[4]
应用场景和实验设计
探究目的蛋白和核酸之间的互作关系,这里的核酸可以是mRNA,也可以是非编码RNA,如circRNA,lncRNA,miRNA等。
样本类型:一般为细胞裂解液。
实验分组:和ChIP类似需要相应的对照。
① 实验内对照:即Input RNA,作为RIP实验的内对照。
② 阳性抗体对照:常用组蛋白抗体作为阳性对照,比如sn RNP70。
③ 阴性抗体对照:常用IgG作为阴性对照组,也可以选择目的蛋白抗体宿主的血清蛋白
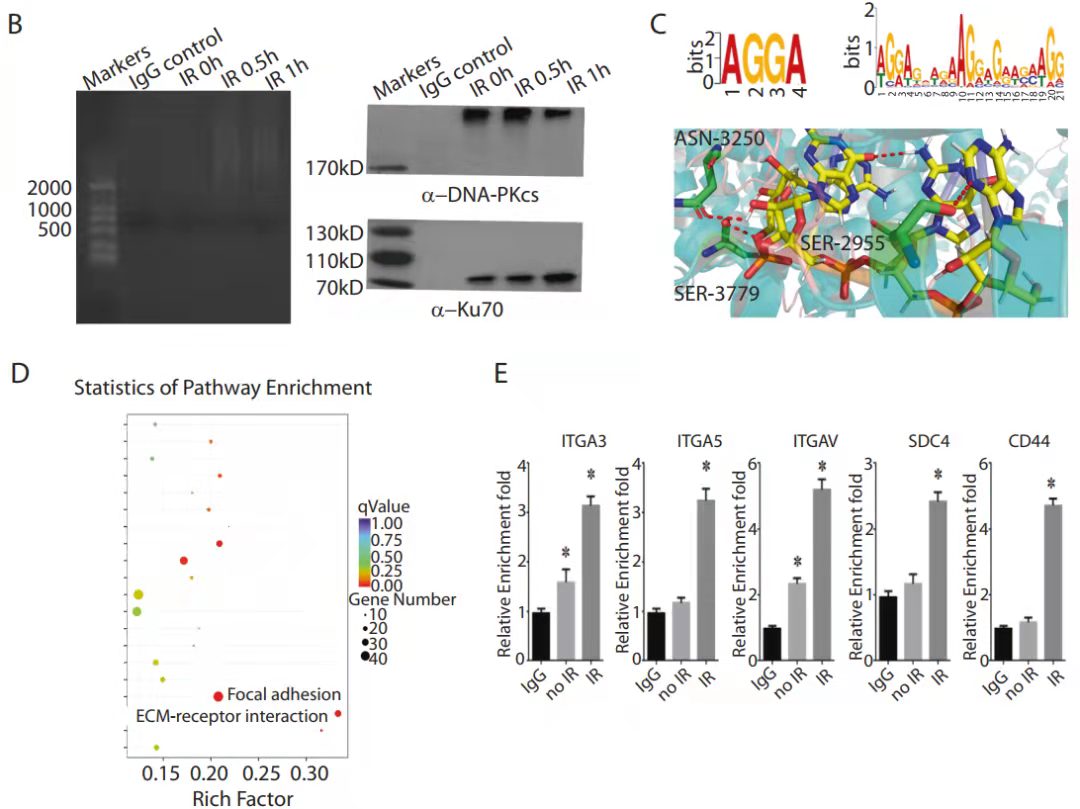
RIP-seq和RIP- PCR结果展示[5]
结果说明:该实验是RIP-seq后通过生信分析,挑选了待验证的基因做了qPCR验证。其中B图是实验组和对照组的IP质控数据,左图是富集后的RNA质量检测,右图验证了抗体的有效性;C-D是DNA测序后的生信分析,分别是motif和对所富集基因的KEGG分析。E检测在有无IR处理条件下,DNA-PKcs和ITGA3、ITGA5 、ITGAV、SDC4、CD44的互作。
实验原理
RNA pulldown也是研究蛋白和RNA互作关系的一种技术手段,与RIP不同的是,此实验原理是利用体外转录法合成RNA,同时标记上生物素作为探针,通过与链霉亲和素标记的磁珠结合从而实现富集细胞裂解液中蛋白的过程,经常和RIP一起使用,互相验证实验结果的可靠性。对于富集后的产物,可以做质谱鉴定和RNA结合的未知蛋白有哪些; 也可以通过Western blot鉴定是否和某已知蛋白存在互作。
应用场景和实验设计
样本类型:一般为细胞裂解液。
实验分组:目的基因探针组(sense)、阴性探针组(anti-sense)
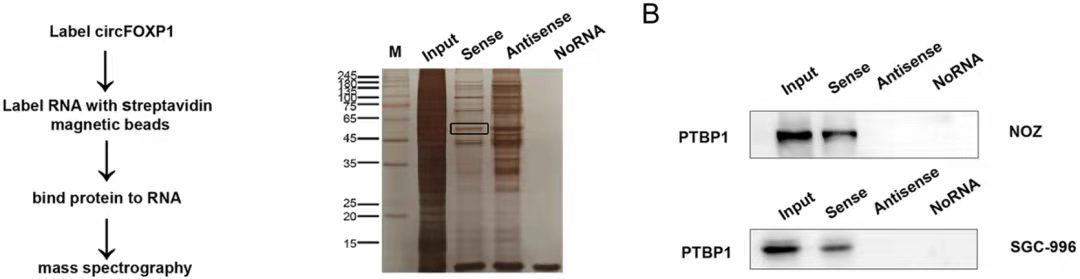
RNA-pulldown-WB图示[6]
结果说明:该实验结果一般会提供一个蛋白银染的数据,用以说明目的探针组和阴性探针组富集条带的差异性,然后选择富集的条带做质谱分析以获得潜在的和RNA存在互作的蛋白信息。
实验原理
将目的基因转录调控原件如某转录因子构建到带有荧光素酶(Firefly luciferase)的表达载体构建成报告基因质粒。将报告基因质粒转染细胞如293T,之后裂解细胞并加入底物荧光素(luciferin),荧光素酶可催化luciferin发出荧光(最强波长在560nm左右)。检测得到的荧光值高低可以判断不同处理组对该转录调控的影响水平。由于会转入Renilla luciferase的报告基因质粒作为内参(最强波长在465nm左右),因此称为双荧光素酶报告基因系统。
应用场景和实验设计
样本类型:一般是在293T细胞中验证。
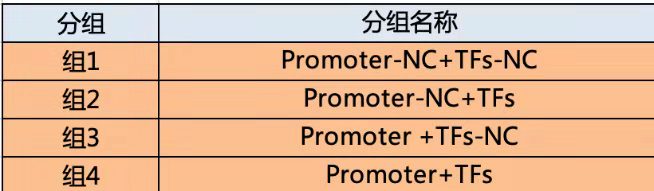
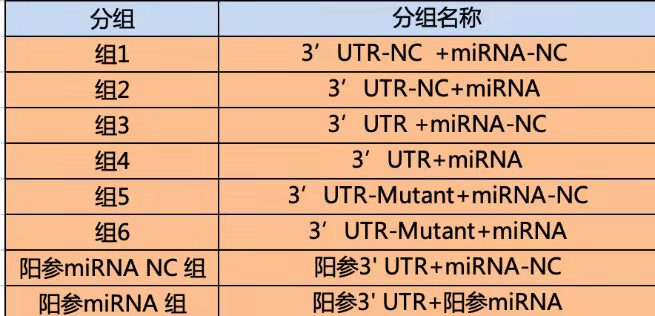
实验分组:研究转录因子和启动子一般分为4组,研究3‘UTR- miRNA的一般有6组。
启动子和转录因子:
miRNA和靶基因:
这些蛋白-核酸互作在实际应用中,可以通过哪些组合设计,高效提升机制探究的效率呢?通过一篇文献和大家简要说明哪些互作实验可以联动,用以解释哪些重要科学问题。


meRIP、RIP-seq鉴定目的基因KIAA1429的下游机制基因GATA3[7]
作者前期在临床样本中发现了影响肝癌患者生存预后以及影响肝癌细胞增殖、转移(细胞和动物水平)的重要基因KIAA1429。该基因是m6A writers,因此作者首先检测敲减KIAA1429后细胞中m6A修饰的变化,发现敲减此基因后,RNA的m6A修饰下降(a-b,e),通过和RIP-seq、RNA-seq测序数据联合分析,发现7个交集基因,并且通过RIP-seq数据发现,其中GATA3与KIAA1429作用更强。
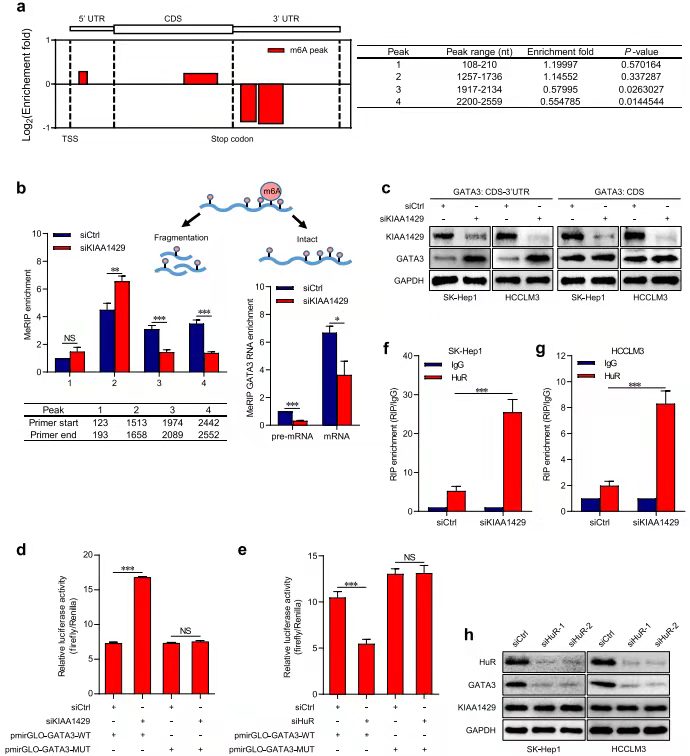
KIAA1429通过调控GATA3 的3 ' UTR m6A修饰而影响其表达[7]

病毒实验帮
公众号底部菜单栏【新功能】上线!
免费在线学习《国自然热点研究》、《数据库及软件操作教程》
一键下载《病毒使用手册》、《高分文献》
还有不定时的送新书、抽奖活动,赶紧来关注一波吧
MeRIP-seq结果显示敲减KIAA1429细胞中的GATA33 基因3' UTR区域的m6A高度富集(a-b),并且通过WB看出下调KIAA1429的CDS-3’UTR后,GATA3表达上调,而在敲减CDS区未发现此现象(c)。RIP-PCR结果显示敲减KIAA1429后RNA结合蛋白HuR上调。然后通过Luciferase实验验证此调控方式。进而得出结论:调控GATA3表达的途径是kiaa1429介导的m6A修饰GATA3 3 ' UTR。
【参考文献】
1.Chen, X., et al., Bcl-3 regulates TGFbeta signaling by stabilizing Smad3 during breast cancer pulmonary metastasis. Cell Death Dis, 2016. 7(12): p. e2508.
2.Nakato, R. and T. Sakata, Methods for ChIP-seq analysis: A practical workflow and advanced applications. Methods, 2021. 187: p. 44-53.
3.Cao, L.Q., et al., Exosomal miR-21 regulates the TETs/PTENp1/PTEN pathway to promote hepatocellular carcinoma growth. Mol Cancer, 2019. 18(1): p. 148.
4.Qi, Y., et al., MALAT1 long ncRNA promotes gastric cancer metastasis by suppressing PCDH10. Oncotarget, 2016. 7(11): p. 12693-703.
5.Song, Z., et al., Genome-wide identification of DNA-PKcs-associated RNAs by RIP-Seq. Signal Transduct Target Ther, 2019. 4: p. 22.
6. Wang, S., et al., Circular RNA FOXP1 promotes tumor progression and Warburg effect in gallbladder cancer by regulating PKLR expression. Mol Cancer, 2019. 18(1): p. 145.
7. Lan, T., et al., KIAA1429 contributes to liver cancer progression through N6-methyladenosine-dependent post-transcriptional modification of GATA3. Mol Cancer, 2019. 18(1): p. 186.
